[23’ NIPS] LLaVA-Med: Training a Large Language-and-Vision Assistant for Biomedicine in One Day
카테고리: Medical
태그: Dataset Multimodal
🔍 Abstract
ChatGPT의 출시 이후 2022년부터 이를 Biomedical Domain에 적용하려는 시도가 계속되고 있었다. 그러나, 당시에는 Image Input을 처리할 수 있는 Multimodal Chatbot은 Visual Med-Alpaca를 제외하고는 없었다. 그러나 Visual Med-Alpaca도 Visual Feature를 온전히 사용하는 것이 아니라 Additional Module을 이용해 Caption/Description으로 변경하여 Text Input으로 처리하는 것이었기 때문에 진정한 의미의 Multimodal Chatbot이라고 할 수 없었다.
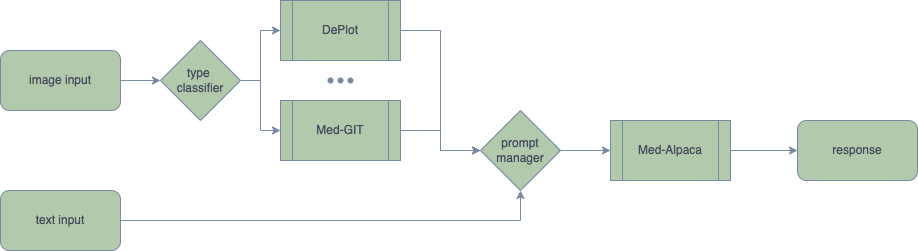
Visual Med-Alpaca의 구조 (출처: Visual Med-Alpaca)
따라서 이 논문에서는 진정한 의미의 Multimodal Biomedical Chatbot인 LLaVA-Med(Large Language and Vision Assistant for BioMedicine)를 제안한다. Key Idea는 다음과 같다.
- PMC(PubMed Central) 데이터로부터 Figure-Caption Dataset을 얻는다.
- 이러한 Dataset과 GPT-4를 이용해 open-ended instruction-following data를 생성한다.
- Curriculum learning method, 즉 Alignment 후 Instruction Tuning을 시행하는 방식으로 LLaVA를 학습한다.
이 논문은 NeurIPS 2023 Track Datasets and Benchmarks에서 Spotlight를 받았다. 그만큼 LLaVA-Med 논문에서 만든 데이터셋과 평가 방법이 높은 가치가 있다고 평가한 것인데, 따라서 Dataset과 Evaluation Method를 집중해서 살펴보도록 하자.
1. Method
LLaVA-Med는 위와 같은 Curriculum Learning 방식으로 학습된다. 즉, (1) Medical Concept Alignment과 (2) Medical Instruction Tuning, 그리고 Task에 따라 추가적인 (3) Downstream Task Fine-tuning으로 이루어진다. 기존의 LLaVA와 동일하게, Alignment 과정에서는 Projection Matrix만 훈련하고, Instruction Tuning 과정에서는 Projection Matrix와 LLM을 훈련한다. 각각의 데이터셋을 어떻게 구축했는지가 중요하다. 순서대로 알아보자.
1.1. Medical Concept Alignment
Biomedical Concept Alignment Data는 PMC-15M, 즉 PubMed-Central Dataset으로부터 600K의 image-text pair를 얻어 훈련하였다. 이때 이들은 figure-caption data이다. 이때 alignment를 위해 아래와 같은 instruction-following data를 생성하였다.
1.2. Medical Instruction Tuning
Biomedical Instruction-Tuning Data를 생성할 때는 더 깊이 있는 이해를 위해 figure에 대해 caption 뿐 아니라 figure를 언급한 논문 내의 문장들(IM, In-line Mention)을 추가하였다. 이들을 GPT-4를 통해 open-ended instruction-following data로 변환하였다. LLaVA와 같이, GPT-4는 image-blind model이기 때문에 text input이 gold-standard라고 가정한다.
이때 Instruction Tuning Data의 중요성을 확인하기 위해 60K-IM(In-line Mention을 추가한 60K 데이터), 60K(기존 Caption만을 이용해 생성한 60K 데이터), 10K로 학습한 경우를 비교하였고, 이로부터 데이터셋의 크기와 IM의 중요성을 알 수 있었다.
이러한 Instruction-Tuning Data에 대한 통계는 다음과 같다. Image data의 경우 CXR, MRI, CT, Histology, Gross Pathology 데이터를 모두 사용하였으며, MRI 및 CT 데이터의 경우 병리적으로 핵심적인 위치를 Slice로 얻은 2D 데이터를 사용한 것으로 보인다.
2. Experiments
2.1. Main Results
LLaVA-Med의 Multimodal Biomedical Chatbot 능력을 검증하기 위해 3개의 biomedical VQA dataset을 이용하였다. 이때 gold standard로는 text-input GPT-4를 이용하여 비교하였다.
결과는 위와 같이 LLaVA-Med가 SOTA의 성능을 보였다. 기존 방법들은 Open-question도 classification task로 처리하여 generalizability가 떨어지는 반면, LLaVA-Med는 Open-ended output을 얻을 수 있어 더욱 generalizable하다.
2.2. Ablation Study
Ablation study로부터 몇 가지 중요한 점을 알 수 있다. (1) BioMedCLIP과 같이 Domain-specific vision encoder를 사용하는 것이 성능 향상에 도움이 된다. (2) Pre-training (Alignment) 만으로는 성능을 얻을 수 없고, Instruction Tuning이 중요하다. (3) Fine-tuning을 충분히 하면 성능 향상에 큰 도움이 된다. 다른 말로 하면 아직 zero-shot ability가 썩 좋지 않다고도 볼 수 있다.
💡 Summary
LLaVA-Med는 방법론적으로는 LLaVA와 크게 다르지 않다. 그러나 거의 최초로 이러한 데이터셋을 구축하고, 성공적인 Generative Biomedical LM을 만들었다는 점에서 높은 가치가 있다. 이러한 방법론은 다른 Domain에도 동일하게 적용할 수 있기에 활용성이 높다.
댓글 남기기